2. 山东省林业科学研究院林木遗传育种重点实验室, 济南, 250014
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2013 年, 第 11 卷, 第 4 篇 doi: 10.5376/mpb.cn.2013.11.0004
收稿日期: 2013年01月11日 接受日期: 2013年03月13日 发表日期: 2013年04月23日
引用格式(中文):
宋伟栓等, 2013, 国槐SRAP-PCR反应体系的建立与优化, 分子植物育种(online), 11(4): 1019-1024 (doi: 10.5376/mpb.cn.2013.11.0004)
引用格式(英文):
Song et al., 2013, Establishment and Optimization of SRAP-PCR Reaction System in Sophora japonica L., Fenzi Zhiwu Yuzhong (online) (Molecular Plant Breeding), 11(4): 1019-1024 (doi: 10.5376/mpb.cn.2013.11.0004)
本实验以国槐叶片DNA为模板,利用SRAP(相关序列多态性)分子标记技术,采用L16(45)正交设计和单因子设计相结合的方法,对国槐SRAP-PCR反应体系中主要因素(DNA、Mg2+、dNTPs、primer、ExTaqDNA聚合酶)进行优化筛选,得出如下结论:各因素不同水平对PCR反应结果都有显著影响,其中ExTaq 酶量影响最大;筛选出各反应因素的最佳水平,确立SRAP-PCR反应的最佳体系(20 μL)为:Mg2+浓度 2.0 mmol/L,模板DNA 80 ng,dNTPs 2.5 mmol/L,引物0.6 μmol/L,ExTaqDNA聚合酶0.75 U,10×PCR buffer2.5 mmol/L;应用该优化体系对退火温度进行了筛选,得出50℃为最佳退火温度。利用该优化体系,从100对SRAP 引物组合中筛选出6对扩增条带清晰,多态性高的引物组合,多态性条带比率高达88.55%。该优化体系的建立为下一步进行古国槐遗传多样性分析,新品种的选育和种质资源的保存利用等奠定了基础。
国槐(Sophora japonica L.)是蝶形花科(Papilionoideae)槐属(Sophora)植物,又叫槐树、巨槐、细叶槐、中国槐、槐米树、守宫槐等,是我国特有的集材用、药用、食用、观赏于一体的树种,在园林绿化中占有重要地位(火树华, 1990, 中国林业出版社, 221-224)。目前,有关国槐繁殖技术(袁秀云等, 2007)和遗传转化方面的研究比较多(张晓英等, 2009),国槐种子不同性状的表型变异及规律研究也有报道(孙荣喜, 2011),但是,国槐分子技术应用上比较薄弱,因此,开展新型标记研究古国槐遗传多样性,为国槐遗传资源评价、保存和利用提供依据,对促进古国槐分子育种进程具有重要意义。
相关序列扩增多态性(sequence-related amplified polymorphism, SRAP)是新型的PCR分子标记技术,于2001年由美国加州大学蔬菜作物系Li与Quiros博士在芸苔属植物上开发而来(Li and Qurios, 2001),该标记多态性高、重复性好、在基因组中分布均匀、引物通用性强等(柳李旺等, 2004, 南京农业大学学报, 26(5): 777-781);SRAP正反相引物可以两两随机搭配组对,用少数引物得到多对引物组合,这样使得引物的使用效率提高了,引物的合成成本也降低了。目前该标记已被成功应用于遗传图谱构建(林忠旭等, 2003, 科学通报, 48(15): 1676-1679)、杂种纯度鉴定(文雁成等, 2006)、遗传多样性分析(陈伦林等, 2008)等方面研究工作。
利用SRAP分子标记进行遗传多样性分析时,PCR体系非常重要,何正文等(1998)较早提出了正交设计优化PCR体系的方法,该方法综合考查了PCR反应体系中各因素及其互作影响,较快得到理想的试验结果,提高效率。本实验采用L16(45)正交设计和单因子相结合的试验,从Mg2+浓度、DNA模板浓度、dNTPs浓度、引物浓度、ExTaq酶浓度以及退火温度等几个方面进行国槐SRAP-PCR主要反应条件的探讨,最后确立最优反应系统,为今后进行古国槐遗传多样性分析及育种工作奠定良好的基础。
1结果与分析
1.1 SRAP-PCR正交实验结果分析
国槐L16(45)正交因素水平反应见表2,电泳实验结果如图1。根据遗传多样性分析要求,对PCR扩增结果依据电泳谱带的强弱及多少,从高到低依次打分,将16个处理结果划分为16个评分等级以便统计分析(何正文等, 1998)。这种打分的方法,主观性比较强,为了使结果更客观,我们根据扩增条带数量丰富、清晰度高、背景低、特异性条带和非特异性条带之间的差异明显的处理,同时参考条带亮度和背景清晰程度为每个处理赋值,进行数据分析。实验重复两次,16个处理赋值数分别为1, 5, 10, 7, 8, 4, 9, 3, 16, 11, 15, 12, 14, 13, 2, 6和3, 5, 8, 9, 12, 11, 13, 2, 16, 15, 14, 10, 6, 7, 1, 4。将上述两次16个处理的评分结果用SPSSV18.0进行方差分析,分析结果见表1。F值反映了各因素对PCR体系显著性影响情况,影响随F值的增大而增大,反之越小,由表1知,正交设计各因素反应水平对反应体系的影响为ExTaq酶>引物>DNA模版>Mg2+>dNTPs。
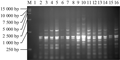 图1 SRAP-PCR正交实验设计PCR电泳 Figure 1 Electrophoresis result of SRAP-PCR by orthoronal design |
 表1 SRAP-PCR反应各因素间方差分析表 Table 1 Variance analysis for the factors of SRAP-PCR reaction |
1.2单因子试验结果分析
1.2.1 Mg2+浓度对PCR扩增效果的影响
Mg2+浓度较高时可以增加产量,但也会降低反应的忠实性,还会影响ExTaqDNA聚合酶的活性,直接影响产量(陈万胜等, 2008; 王燕青和季孔庶, 2009)。Mg2+能与反应液中的引物、模板及dNTPs相结合,使引物与模板的结合效率受到影响,还影响产物特异性及形成引物二聚体等(孟宪婷等, 2009),试验选取了(0.2 mmol/L, 0.5 mmol/L, 1.0 mmol/L, 1.5 mmol/L, 2.0 mmol/L, 2.5 mmol/L)浓度的Mg2+进行了PCR扩增,结果显示:Mg2+为2.0 mmol/L时扩增条带清晰,且出现了特异性条带,当浓度过高时,谱带又变弱,最终确定Mg2+浓度为2.0 mmol/L,如图2中5泳道。
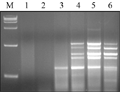 图2 不同Mg2+浓度的SRAP反应结果 Figure 2 The SRAP profile with different Mg2+ concentrations |
1.2.2 DNA浓度对PCR扩增效果的影响
DNA模板的浓度和质量,是PCR成败的关键因素之一,太少时没有扩增条带,太多时则会出现非特异性条带(彭飒等, 2006, 第二军医大学学报, 27(5): 544-547)。试验选用不同DNA模板浓度(40 ng, 60 ng, 80 ng, 100 ng, 120 ng, 140 ng)进行扩增,结果显示:DNA模版在80~120 ng之间没有明显的差异,考虑经济因素,确定80 ng模板量为宜,如图3中3泳道。
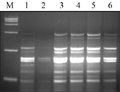 图3不同DNA浓度的SRAP反应结果 Figure 3 The SRAP profile with different DNA concentrations |
1.2.3 dNTPs浓度对PCR扩增效果的影响
dNTPs与扩增效率密切相关,过高时与ExTaqDNA酶竞争Mg2+,过低时会使扩增产物单链化,两者都对扩增不利(孟宪婷等, 2009)。试验选取(1 mmol/L, 1.5 mmol/L, 2.0 mmol/L, 2.5 mmol/L, 3.0 mmol/L, 3.5 mmol/L)的dNTPs浓度进行扩增,结果显示:1.5 mmol/L、0.20 mmol/L浓度时效果较好,综合考虑,确定dNTPs最适浓度为2.0 mmol/L,如图4中3泳道。
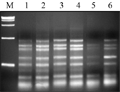 图4 不同dNTPs浓度的SRAP反应结果 Figure 4 The SRAP profile with different dNTPs concentrations |
1.2.4引物浓度对PCR扩增效果的影响
引物浓度能够影响扩增特异性,过高会加剧非特异性扩增和错配的发生;引物浓度过低会使PCR扩增效率降低(任羽等, 2004),所以选择合适的引物浓度很重要。试验选取(0.4 μmol/L, 0.6 μmol/L, 0.8 μmol/L, 1 μmol/L, 1.2 μmol/L, 1.4 μmol/L)的引物浓度,研究结果显示:伴随引物浓度不断加大,扩增的条带变得逐渐清晰、丰富,过高时条带又变弱,在0.6 μmol/L时出现特异性条带,在0.6-0.8 μmol/L之间没有差异,最终确定引物浓度为0.6 μmol/L,如图5中2泳道。
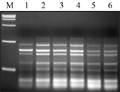 图5不同引物浓度的SRAP反应结果 Figure 5 The SRAP profile with different primer concentrations |
1.2.5 ExTaq酶浓度对PCR扩增效果的影响
酶用量是PCR重要影响因素之一,ExTaqDNA聚合酶浓度过低时无扩增,浓度过高又会引起非特异性扩增。实验设置6个ExTaq酶浓度(0.25 U, 0.5 U, 0.75 U, 1.0 U, 1.25 U, 1.5 U),结果显示:酶浓度在0.75 U、1.0 U时扩增条带较多,用量为1.25 U时,产物条带出现扩散,综合考虑,ExTaq酶浓度确定为0.75 U,如图6中3泳道。
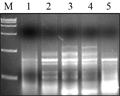 图6不同Extaq酶浓度的SRAP反应结果 Figure 6 The SRAP profile with different Extaq DNA polymorases concentrations |
1.2.6退火温度对PCR扩增效果的影响
退火温度是影响PCR特异性的重要因素,根据引物的长短、碱基和浓度而定。较低的退火温度可以使引物与模板发生结合,但易产生非特异性条带;过高又抑制引物与模板的结合。试验在PCR仪自动生成的温度梯度中选择6个温度扩增(48.1℃, 49.4℃, 50.7℃, 52.6℃, 54.1℃, 55.5℃),结果表明:温度为48℃或高于55℃时,PCR产物量较低,条带亮度弱,而在50℃和52℃温度时扩增稳定。因此,确定退火温度为50℃如图7中3泳道。
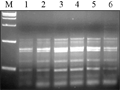 图7不同退火温度SRAP反应结果 Figure 7 The SRAP profile with different annealing temperature concentrations |
1.3优化反应体系的验证
使用初步筛选的引物组合Me11/Em5,对16份不同种源的国槐进行扩增,验证上述优化体系,结果显示(图8),每份样品均扩增出DNA片段,条带清晰,说明该体系稳定,适合国槐SRAP-PCR反应。
|
图8 引物ME11-EM5对国槐16个样品的SRAP扩增电泳结果 Figure 8 SRAP profiles amplified among 16 grape cultivars with primer pair M11-E5 |
2结论与讨论
SRAP被成功应用于多种植物的遗传多样性分析、相关基因克隆、遗传图谱构建以及重要的性状标记等,如红松(Chen et al., 2010)、柿树(Guo and Luo, 2006)甜瓜(王建设等, 2007)等,基于PCR反应的SRAP标记,反应体系中任何一种因素的改变都会影响扩增结果的变化,各组分间相互作用的浓度较难确定。本实验采用单因素完全设计和正交实验设计相结合的方法,共同探讨了国槐SRAP-PCR中各影响因子的规律及最佳浓度范围,正交实验结果显示以9泳道最佳(图1),其主要反应因素浓度为:Mg2+ 1.5 mmol/L,DNA 80 ng,dNTPs 2.5 mmol/L,引物0.8 μmol/L,ExTaqDNA 1.0 U,单因子实验结果显示:Mg2+ 1.5 mmol/L,DNA 80 ng,dNTPs 2.5 mmol/L,引物0.8 μmol/L,ExTaqDNA 0.75 U。这两种实验结果都表明DNA 80 ng,dNTPs 2.5 mmol/L时最佳。通过多次多水平实验证明,Mg2+浓度2.0 mmol/L,引物0.6 μmol/L,ExTaqDNA聚合酶0.75 U时,此反应体系扩增条带更清晰,重复性和稳定性较好,所以,最终建立的SRAP体系(20 μL)为:Mg2+浓度2.0 mmol/L,模板DNA 80 ng,dNTPs 2.5 mmol/L,引物0.6 μmol/L,ExTaqDNA聚合酶1.0 U,10×PCR Buffer 2.5 mmol/L;退火温度为50℃时最佳。利用该体系进一步进行国槐遗传多样性分析,对不同地理种源的国槐进行遗传分化度和亲缘关系的研究,为国槐分子辅助育种,种质资源的保存和利用提供一定的理论基础。
3材料与方法
3.1材料与试剂
供试材料分别来自山东潍坊、淄博、滨州、泰安、东营等地的野生古国槐资源,由2012年3根据山东省古树索引资料,搜集到古国槐无性系,嫁接于山东省林业科学研究院苗圃基地。整个实验以古国槐无性系叶片为材料。
试验所用ExTaqDNA聚合酶,Mg2+,10×PCR Buffer,dNTPs等试剂均购自大连TaKaRa公司;SRAP引物采用的组合参考文献(Li and Qurios, 2001; Ferriol et al., 2003),由上海博尚生物工程技术有限公司合成。
3.2实验方法
3.2.1 DNA提取与检测
采用改良CTAB法提取样品材料中的DNA (Fjellstrom et al., 1994)。用0.8%的琼脂糖凝胶电泳对提取样品DNA进行初步检测后,在紫外分光光度计下对其浓度和纯度进行定量检测,并将DNA稀释到约50 ng/μL,于-20℃条件下保存。
3.2.2 PCR反应程序的确定
参考文献(Li and Qurios, 2001)的扩增程序:94℃预变性5 min;94℃变性1 min-35℃退火1 min-72℃延伸1 min,5个循环;94℃变性1 min-50℃退火1 min-72℃延伸1 min,进行35个循环;最后72℃延伸10 min,4℃保存。
3.2.3 PCR反应因素水平的确定
为了确立SRAP反应体系(20 μL)中主要因素:ExTaq酶、引物、DNA模版、Mg2+和dNTPs。的最佳水平,该实验采用L16(45)正交设计和单因子设计相结合的方法,各因素水平见表2。引物对序列为ME11:TGAGTCCAAACCGGATA,EM5:TGAGTCCTTTCCGGTAA。
|
表2 SRAP-PCR反应因素水平 Table 2 factors levels of PCR reaction |
3.2.4 SRAP优化体系的验证
利用优化好的SRAP体系,多不同国槐样品进行扩增,引物组合同上(3.2.3),PCR产物用含EB 0.5 mg/L琼脂糖凝胶(1.5%)电泳检测紫外光下GeneGenius观察成像。
作者贡献
宋伟栓是本研究的实验设计和实验研究的执行人;庞彩红、李双云及李丽完成数据分析;杨克强参与实验设计,试验结果分析;夏阳是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由山东省农业生物资源创新利用研究课题(2001157)资助。
参考文献
Chen L.L., Zou X.Y., Li S.Y., Zou X.F., Zhang J.M., and Song L.Q., 2008, Analysis the diversity of rapeseed by SSR and SRAP with emphasis on the diference ofthis two kinds of markers, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 6(3): 5l1-516 (陈伦林, 邹小云, 李书宇, 邹晓芬, 张建模, 宋来强, 2008, SSR和SRAP标记揭示甘蓝型油菜遗传多样性的差异分析, 分子植物育种, 6(3): 511-516)
Chen M.M., Feng F.J., Sui X., Li M,H., Zhao D., and Han S., 2010, Construction of a frame map for Pinus koraiensis Sieb. et Zucc.using SRAP, SSR and ISSR markers, Trees, (24): 685-693
http://dx.doi.org/10.1007/s00468-010-0438-5
Chen W.S., Wang Y.Y., Luo C.G., Yang A.G., Jiang C.H., and Fan J.Y., 2008, Optimization for SRAP-PCR system of tobacoo based on orthogonal design, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 6(1): 177-182 (陈万胜, 王元英, 罗成刚, 杨爱国, 蒋彩虹, 范静苑, 2008, 利用正交设计优化烟草SRAP反应体系, 分子植物育种, 6(1): 177-182)
Ferriol M., Pico B., and Nuez F., 2003, Genetic diversity of a germplasm collection of Cucurbita pepo using SRAP and AFLP markers, Theor. Appl. Genet., 107(2): 271-282
http://dx.doi.org/10.1007/s00122-003-1242-z
PMid:12845442
Fjellstrom R.G., Parfitt D.E., and McGranahan G.H., 1994, Genetic relationships and characterization of Persian walnut (Juglans regia L.) cultivars using restriction fragment length polymorphisms (RFLPs), Journal of the American Society for Horticultural Science, 119(4): 833-839
Guo D.L., and Luo Z.R., 2006, Genetic relationships of some PCNA persimmons (Diospyros kaki Thunb.) from China and Japan revealed by SRAP analysis, Genetic Resources and Crop Evolution, (53): 1597-1603
http://dx.doi.org/10.1007/s10722-005-8717-5
He Z.W., Liu Y.S., Chen L.H., Cao M.H., and Xia J.H., 1998, Orthogonal design-direct analysis for PCR optimization, Hunan Yike Daxue Xuebao (Bulletin of Hunan Medical University), 23(4): 403-404 (何正文, 刘运生, 陈立华, 曹美鸿, 夏家辉, 1998, 正交设计直观分析法优化PCR条件, 湖南医科大学学报, 23(4): 403-404)
Li G., and Qurios C.F., 2001, Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica, Theor Appl Genet, 103: 455-461
http://dx.doi.org/10.1007/s001220100570
Meng X.T., Chi D.F., Wang X.H., and Xia D., 2009, Optimization of SRAP reaction system in Fraxinus mandshu rica, Dongbei Linye Daxue Xuebao (Journal of Northeast Forestry), 37(11): 13-16 (孟宪婷, 迟德富, 王秀华, 夏德安, 2009, 水曲柳SRAP分子标记反应系统的优化, 东北林业大学学报, 37(11): 13-16)
Ren Y., Wang D.Y., Zhang Y.D., Li Y., and Wang H.M., 2004, Optimization of SRAP-PCR in hot pepper (Capsicum annuItm L), Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 2(5): 689-693 (任羽, 王得元, 张银东, 李颖, 王恒明, 2004, 辣椒SRAP-PCR反应体系的建立与优化, 分子植物育种, 2(5): 689-693)
Sun R.X., 2011, Seed morphological variation of different sources and molecular identification of cultivars /clones of Sophora japonica L. by SRAP marker, Thesis for M.S., Chinese Academy of Forestry, Supervisor: Zheng Y.Q., and Zhang C.H., pp.1-2 (孙荣喜, 2011, 国槐种子形态变异与品种/无性系SRAP分子识别研究, 硕士学位论文, 中国林业科学研究院, 导师: 郑勇奇, 张川红, pp.1-2)
Wang J.S., Yao J.C., Liu L., Wang Y.J., and Li W., 2007, Construction of a molecular genetic map for melon (Cucumis melo L.) based on SRAP, Yuanyi Xuebao (Acta Horticulturae Sinica), 34(1): 135-140 (王建设, 姚建春, 刘玲, 王永健, 李唯, 2007, 利用中国香瓜与哈密瓜的F2群体构建SRAP连锁遗传图谱, 园艺学报, 34(1): 135-140)
Wang Y.Q., and Ji K.S., 2009, Optimization for SRAP-PCR system of Paeonia suffruticosa based on orthogonal design, 7(1): 199-203 (王燕青, 季孔庶, 2009, 利用正交设计优化牡丹SRAP-PCR反应体系, 分子植物育种,7(1): 199-203)
Wen Y.C., Wang H.Z.,Shen J.X., and Liu G.H., 2006, Comparision of cultivar fingerprints constructed with SRAP and SSR markers in Brassica napus L., Zhongguo Youliao Zuowu Xuebao (Chinese Journal of Oil Crop Sciences), 28(3): 233-239 (文雁成, 王汉中, 沈金雄, 刘贵华, 2006, SRAP和SSR标记构建的甘蓝型油菜品种指纹图谱比较, 中国油料作物学报, 28(3): 233-239)
Yuan X.Y., Zhang X.Y., Ma J., and Sang H.M., 2007, Research on tissue culture and regeneration technology of Sophora japonica, Anhui Nongye Kexue (Journal of Anhui Agricultural Sciences) 35(35): 11418- 11419 (袁秀云, 张仙云, 马杰, 桑慧敏, 2007, 国槐植株再生技术研究, 安徽农业科学,35(35): 11418- 11419)
Zhang X.Y., Gan J., Yin W.L., Zhu Z., and Wang H.F., 2009, An effective transformation system of Sophora japonica, Linye Kexue (Scientia Silvae Sinicae), 45(5): 20-26 (张晓英, 甘敬, 尹伟伦, 朱祯, 王华芳, 2009, 国槐遗传转化体系的优化, 林业科学, 45(5): 20-26)